NanoLuc® Luciferase Technology
NanoLuc® Luciferase Technology
テクノロジー概要
- NanoLuc®とは
-
NanoLuc®(Nluc)は深海エビ(トゲオキヒオドシエビ[Oplophorus gracilirostris])由来のルシフェラーゼです。触媒活性のある19kDaのサブユニットを改変し、さらに本来の基質であるCoelenterazineをもとに作製したFurimazineという新しい基質を用いることで発光強度と安定性を高めました。発光レポーターとして最適なパフォーマンスを発揮するために作り出されたルシフェラーゼです。
参考文献 Hall, M.P., et al. (2012) ACS Chem Biol 7:1848
非常に高い発光レベル
- 高感度により正確な生体内イベントを検出可能
- ホタルルシフェラーゼ (luc2)よりも80倍~240倍のシグナル(細胞内発現)
非常に小さいレポーター分子(19kD)
- ホタル/ウミシイタケルシフェラーゼやGFPよりも小さい分子量
- 遺伝子やタンパク質サイズに制約がある用途にも対応(例:ウイルスパッケージング、タンパク質融合)
長時間発光
- すべての処理スケールに適応
- 容易に自動化へ対応(例:HTS)
高い安定性
- 幅広い実験条件に適応(温度、pH 他)
- 化合物ライブラリースクリーニングでの疑陽性を低減
多用性
- 標準的な細胞溶解アッセイ
- 分泌アッセイ(細胞非溶解)
- タンパク質安定性を変えた細胞内アッセイ
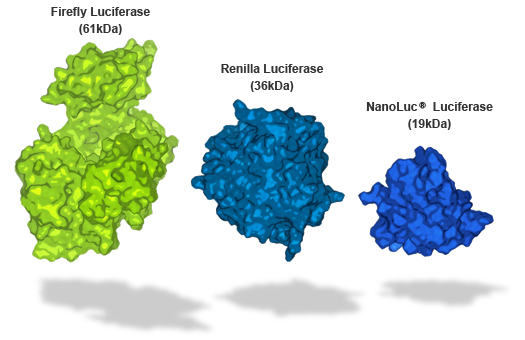
ルシフェラーゼ分子の相対的なサイズ
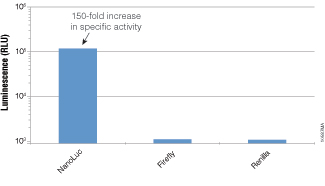
種類ルシフェラーゼ酵素(それぞれ50 attomoles)からの発光測定
ホタルルシフェラーゼはONE-Glo™ Luciferase Assay Reagent、ウミシイタケルシフェラーゼはRenilla-Glo® Luciferase Assay Reagent、NanoLuc® ルシフェラーゼはNano-Glo® Luciferase Assay Reagent をそれぞれ用いてGloMax® Multi Microplate Luminometerで測定した。NanoLuc® ルシフェラーゼは他のルシフェラーゼよりも約150倍の比活性を示した。

96ウェルプレート内のHEK293をiPhoneで撮影(1ウェル細胞50,000個)
- NanoBiT®とは 〜生細胞でのタンパク質間相互作用検出法〜
-
NanoLuc® ルシフェラーゼ開発後、NanoLuc® ルシフェラーゼをベースにした2つの断片で細胞内でのタンパク質相互作用検出を可能にするNanoLuc® 2分子テクノロジー(NanoBiT®:NanoLuc® Binary Technology)を開発しました。NanoLuc® ルシフェラーゼの大きな断片であるLarge BiT (LgBiT)[18 KDa] と相補性を持つ小さなペプチド断片(11 アミノ酸)をスクリーニングし、最も親和性の低い断片(Kd=190 μM)をSmall BiT(SmBiT)としてNanoBiT® システムに採用しました。
LgBiTおよびSmBiTをそれぞれ標的タンパク質との融合体として発現させ、相互作用が起こると断片の相補性が促進され発光酵素として明るい光を生じます。多くのスプリット系とは対照的に LgBiT:SmBiT の相互作用は可逆的でタンパク質間の迅速な解離も検出できます。
- HiBiTとは 〜もうひとつの2分子相補システム〜
-
NanoBiT® においてLgBiTに対し最も親和性の低いペプチド断片SmBiTが採用された一方、最も高い親和性を有するペプチド断片(Kd=700 pM)をHiBiT と名づけ、発現タンパク質を高感度に検出するための発光タグとして活用することになりました。非常に明るい光により過剰発現させることなく内在発現レベルで目的タンパク質の定量化が可能であるため、CRISPR 等のゲノム編集技術と組み合わせて内在ローカスにこのHiBiT タグを埋め込むことで実質的にあらゆるタンパク質の発現をモニタリングすることができます。
参考文献 Schwinn, M.K., et al. (2017) ACS Chem Biol Oct 23.
HiBiTサイトは こちら
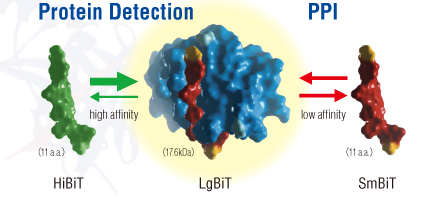
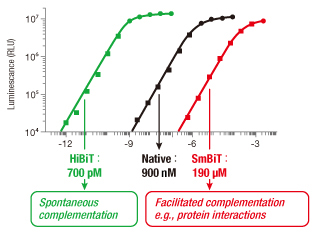 Small BiTまたはHiBiTは、Large BiTとそれぞれ異なる親和性でNanoLuc® を再構成する。
Small BiTまたはHiBiTは、Large BiTとそれぞれ異なる親和性でNanoLuc® を再構成する。
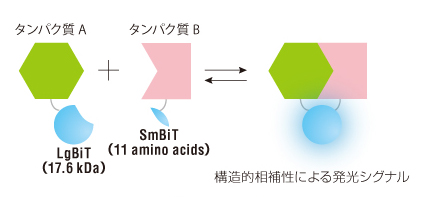 NanoBiT® の構造と再構成によるタンパク質間相互作用の測定原理タンパク質 A と B にそれぞれLarge BiT と Small BiT を融合させて細胞内で発現させ、タンパク質 A と B が相互作用すると NanoBiT® が再構成されて発光を示す。
NanoBiT® の構造と再構成によるタンパク質間相互作用の測定原理タンパク質 A と B にそれぞれLarge BiT と Small BiT を融合させて細胞内で発現させ、タンパク質 A と B が相互作用すると NanoBiT® が再構成されて発光を示す。
NanoLuc®ベクター
- ベクターの特長
-
- Nluc遺伝子のコドンは最適化され、ほとんどの転写因子結合サイト、プロモーターモジュール、スプライスサイトならびにその他の調節配列を除去
- pGL4ベースの構造により既存のプラスミドからの移し変えが容易
- 研究対象となる遺伝子ORFとNanoLuc® が融合したクローンコレクションも有り
- ベクターの種類
-
- レポーターアッセイ用、NanoLuc® との融合タンパク質作製用、シグナル伝達解析用
- 標準型、不安定型、分泌型
- プロモーターの有無(クローニングベクターかコントロールベクターか)
- 安定細胞株作製用のハイグロマイシンマーカーやネオマイシンマーカーの有無
- 第2のレポーター遺伝子(ホタルルシフェラーゼ:Fluc)の有無(あればスクリーニング時の偽陽性排除に役立つ)
28種類の異なるベクターから、実験目的に適したNanoLuc® ルシフェラーゼを選択できます。
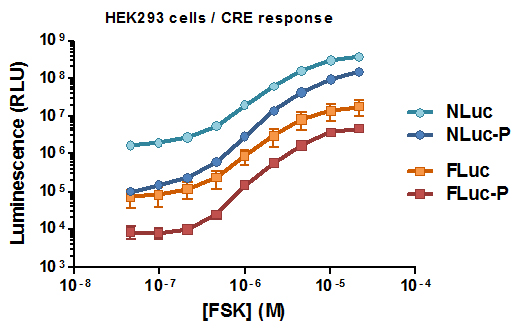
cAMP応答配列 (CRE)制御下のルシフェラーゼを含むコンストラクトをHEK293細胞にトランスフェクションした。フォルスコリンで誘導した後、Nano-Glo® およびONE-Glo® Luciferase Assay Systemsの各試薬とGloMax® 96 Microplate Luminometerを用いてルシフェラーゼ発現を検出した。NanoLuc® ルシフェラーゼは標準型または不安定型ホタルルシフェラーゼよりも明るい発光シグナルを示した。PEST不安定化ドメインはどちらのルシフェラーゼにおいても誘導倍率を向上させ、NanoLuc® を使用した際に最大の応答性が得られた。すべてのケースで誘導によるEC50値はほぼ同様。
| NLuc | FLuc | Nluc-P | Fluc-P | |
|---|---|---|---|---|
| Luminescence (RLV) | 1.7E6 | 6.8E4 | 8.6E4 | 6.0E3 |
| EC50 (μM) | 6 | 6 | 10 | 6 |
| Induction Ratio | 230 | 270 | 1700 | 780 |
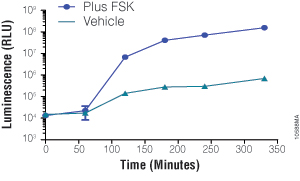
NanoLuc® ルシフェラーゼ (secNluc)のCRE応答性発現を6時間フォルスコリン誘導あるいはDMSOコントロールの細胞で測定した。培地交換をt=0で行った。分泌アッセイフォーマットによりレポーターの活性化キネティクスを細胞溶解することなく測定できた。シグナルはGloMax® 96 Microplate Luminometerを用いて測定した。
アプリケーション
- NanoBRET® とNanoBiT® の特性比較
| 技術 | 実験手法 | 内容 |
|---|---|---|
| NanoLuc® ルシフェラーゼ | NanoBRET® | タンパク質間相互作用検出(生細胞リアルタイムアッセイ) |
| ターゲットエンゲージメント | 分子とタンパク質の相互作用を生細胞リアルタイムモニタリング | |
| バイオセンサー | 細胞内外のシグナリングイベントをBRETベースのバイオセンサーで検出 | |
| イメージング | 細胞または動物個体でのイメージング | |
| タンパク質の安定性 | 細胞内タンパク質の存在量の変動をモニタリング | |
| 遺伝子発現制御 | 感度が格段にアップしたレポーターアッセイ | |
| 発現確認 | PAGE後、NanoLuc® 融合タンパク質のバンドを簡便に可視化 | |
| 2分子相補システム | NanoBiT® | タンパク質間相互作用検出(生細胞リアルタイムアッセイ) |
| HiBiT細胞内タンパク質検出 | 細胞内で発現したHiBiT融合タンパク質の定量(細胞を溶解する) | |
| HiBiT細胞外タンパク質検出 | 細胞膜上に存在、または分泌されたHiBiT融合タンパク質の定量(細胞を生かしたまま測定) | |
| HiBiTブロッティング検出 | ウエスタンのようにメンブレンにブロットしたHiBiT融合タンパク質を検出(抗体不要) |
| 特性 | NanoBRET® | NanoBiT® | |
|---|---|---|---|
| 方式 | 生物発光共鳴 エネルギー転移(BRET) | 発光酵素断片の相補性 | |
| 検出対象 | 近接度 | 直接的な相互作用 | |
| タグの大きさ | 小さい 20 kDa(NanoLuc™)と30 kDa(HaloTag®) |
非常に小さい 11アミノ酸(SmBiT)と<18 kDa(LgBiT) |
|
| データ | 2波長の比率(低 %CV) | RLU(相対発光強度) | |
| 試薬添加回数 | 2 | 1 | |
| 検出機器 | アッセイ | BRET 対応ルミノメーター (適合フィルター要) |
ルミノメーター (フィルター不要) |
| イメージング | 発光イメージングシステム (Olympus LV200 など): 相互作用前後を観察可能 |
発光イメージングシステム (Olympus LV200 など): 複合体のみ観察可能 |
|
| 可逆性 (キネティックアッセイ可) |
◯ | ◯ | |
| 製品形態 | キット(100種類以上)、 クローニングベクター、検出試薬 |
クローニングベクター、 検出試薬 |
|
| NanoBRET® ベクター構築サービス | NanoBiT® ベクター構築サービス | ||
| ライセンス費用(企業を含む) | 試薬の購入のみ | ||
| その他 | HaloTag® 側を使ったプルダウン | ||
サイテーション
NanoBRET
※ 論文タイトルをクリックするとPubMedに移動します| タイトル | 著者 | ジャーナル名 |
|---|---|---|
|
Biochemical analysis of Rabin8, the guanine nucleotide exchange factor for Rab8. |
Wu B, Wang J, Zhao Y, Guo W. | Methods Cell Biol. 2015;130:59-68. |
|
Activation of Rab8 guanine nucleotide exchange factor Rabin8 by ERK1/2 in response to EGF signaling. |
Wang J, Ren J, Wu B, Feng S, Cai G, Tuluc F, Peränen J, Guo W. | Proc Natl Acad Sci U S A. 2015 Jan 6;112(1):148-53. |
|
NanoBRET–A Novel BRET Platform for the Analysis of Protein-Protein Interactions. |
Machleidt T, Woodroofe CC, Schwinn MK, Méndez J, Robers MB, Zimmerman K, Otto P, Daniels DL, Kirkland TA, Wood KV. | ACS Chem Biol. 2015 Aug 21;10(8):1797-804. |
| Bradley WD, Arora S, Busby J, Balasubramanian S, Gehling VS, Nasveschuk CG, Vaswani RG, Yuan CC, Hatton C, Zhao F, Williamson KE, Iyer P, Méndez J, Campbell R, Cantone N, Garapaty-Rao S, Audia JE, Cook AS, Dakin LA, Albrecht BK, Harmange JC, Daniels DL, Cummings RT, Bryant BM, Normant E, Trojer P. | Chem Biol. 2014 Nov 20;21(11):1463-75. | |
|
9H-purine scaffold reveals induced-fit pocket plasticity of the BRD9 bromodomain. |
Picaud S, Strocchia M, Terracciano S, Lauro G, Mendez J, Daniels DL, Riccio R, Bifulco G, Bruno I, Filippakopoulos P. | J Med Chem. 2015 Mar 26;58(6):2718-36. |
|
Target engagement and drug residence time can be observed in living cells with BRET. |
Robers MB, Dart ML, Woodroofe CC, Zimprich CA, Kirkland TA, Machleidt T, Kupcho KR, Levin S, Hartnett JR, Zimmerman K, Niles AL, Ohana RF, Daniels DL, Slater M, Wood MG, Cong M, Cheng YQ, Wood KV. | Nat Commun. 2015 Dec 3;6:10091. |
|
Fluorescence- and bioluminescence-based approaches to study GPCR ligand binding. |
Stoddart LA, White CW, Nguyen K, Hill SJ, Pfleger KD. | Br J Pharmacol. 2016 Oct;173(20):3028-37. |
| Conery AR, Centore RC, Neiss A, Keller PJ, Joshi S, Spillane KL, Sandy P, Hatton C, Pardo E, Zawadzke L, Bommi-Reddy A, Gascoigne KE, Bryant BM, Mertz JA, Sims RJ. | Elife. 2016 Jan 5;5. pii: e10483. | |
|
Deciphering the Cellular Targets of Bioactive Compounds Using a Chloroalkane Capture Tag. |
Ohana RF, Kirkland TA, Woodroofe CC, Levin S, Uyeda HT, Otto P, Hurst R, Robers MB, Zimmerman K, Encell LP, Wood KV. | ACS Chem Biol. 2015 Oct 16;10(10):2316-24. |
|
LP99: Discovery and Synthesis of the First Selective BRD7/9 Bromodomain Inhibitor. |
Clark PG, Vieira LC, Tallant C, Fedorov O, Singleton DC, Rogers CM, Monteiro OP, Bennett JM, Baronio R, Müller S, Daniels DL, Méndez J, Knapp S, Brennan PE, Dixon DJ. | Angew Chem Weinheim Bergstr Ger. 2015 May 18;127(21):6315-6319. |
| Mo XL, Luo Y, Ivanov AA, Su R, Havel JJ, Li Z, Khuri FR, Du Y, Fu H. | J Mol Cell Biol. 2016 Jun;8(3):271-81. | |
|
Generation of a Selective Small Molecule Inhibitor of the CBP/p300 Bromodomain for Leukemia Therapy. |
Picaud S, Fedorov O, Thanasopoulou A, Leonards K, Jones K, Meier J, Olzscha H, Monteiro O, Martin S, Philpott M, Tumber A, Filippakopoulos P, Yapp C, Wells C, Che KH, Bannister A, Robson S, Kumar U, Parr N, Lee K, Lugo D, Jeffrey P, Taylor S, Vecellio ML, Bountra C, Brennan PE, O’Mahony A, Velichko S, Müller S, Hay D, Daniels DL, Urh M, La Thangue NB, Kouzarides T, Prinjha R, Schwaller J, Knapp S. | Cancer Res. 2015 Dec 1;75(23):5106-5119. |
| McGrath JP, Williamson KE, Balasubramanian S, Odate S, Arora S, Hatton C, Edwards TM, O’Brien T, Magnuson S, Stokoe D, Daniels DL, Bryant BM, Trojer P. | Cancer Res. 2016 Apr 1;76(7):1975-88. | |
|
Inhibition of the p53/hDM2 protein-protein interaction by cyclometallated iridium(III) compounds. |
Liu LJ, He B, Miles JA, Wang W, Mao Z, Che WI, Lu JJ, Chen XP, Wilson AJ, Ma DL, Leung CH. | Oncotarget. 2016 Mar 22;7(12):13965-75. |
|
Detection of Antibodies in Blood Plasma Using Bioluminescent Sensor Proteins and a Smartphone. |
Arts R, den Hartog I, Zijlema SE, Thijssen V, van der Beelen SH, Merkx M. | Anal Chem. 2016 Apr 19;88(8):4525-32. |
|
Regulatory T Cell Modulation by CBP/EP300 Bromodomain Inhibition. |
Ghosh S, Taylor A, Chin M, Huang HR, Conery AR, Mertz JA, Salmeron A, Dakle PJ, Mele D, Cote A, Jayaram H, Setser JW, Poy F, Hatzivassiliou G, DeAlmeida-Nagata D, Sandy P, Hatton C, Romero FA, Chiang E, Reimer T, Crawford T, Pardo E, Watson VG, Tsui V, Cochran AG, Zawadzke L, Harmange JC, Audia JE, Bryant BM, Cummings RT, Magnuson SR, Grogan JL, Bellon SF, Albrecht BK, Sims RJ 3rd, Lora JM. | J Biol Chem. 2016 Jun 17;291(25):13014-27. |
|
GSK6853, a Chemical Probe for Inhibition of the BRPF1 Bromodomain. |
Bamborough P, Barnett HA, Becher I, Bird MJ, Chung CW, Craggs PD, Demont EH, Diallo H, Fallon DJ, Gordon LJ, Grandi P, Hobbs CI, Hooper-Greenhill E, Jones EJ, Law RP, Le Gall A, Lugo D, Michon AM, Mitchell DJ, Prinjha RK, Sheppard RJ, Watson AJ, Watson RJ. | ACS Med Chem Lett. 2016 May 9;7(6):552-7. |
| Goyet E, Bouquier N, Ollendorff V, Perroy J. | Sci Rep. 2016 Jun 15;6:28231. | |
| Perez-Perri JI, Dengler VL, Audetat KA, Pandey A, Bonner EA, Urh M, Mendez J, Daniels DL, Wappner P, Galbraith MD, Espinosa JM. | Cell Rep. 2016 Jun 28;16(1):37-47. | |
| Looyenga B, VanOpstall C, Lee Z, Bell J, Lodge E, Wrobel K, Arnoys E, Louters L. | Sci Rep. 2016 Jun 30;6:29130. | |
|
Mutations of Vasopressin Receptor 2 Including Novel L312S Have Differential Effects on Trafficking. |
Tiulpakov A, White CW, Abhayawardana RS, See HB, Chan AS, Seeber RM, Heng JI, Dedov I, Pavlos NJ, Pfleger KD. | Mol Endocrinol. 2016 Aug;30(8):889-904. |
|
Sensitivity and engineered resistance of myeloid leukemia cells to BRD9 inhibition. |
Hohmann AF, Martin LJ, Minder JL, Roe JS, Shi J, Steurer S, Bader G, McConnell D, Pearson M, Gerstberger T, Gottschamel T, Thompson D, Suzuki Y, Koegl M, Vakoc CR. | Nat Chem Biol. 2016 Sep;12(9):672-9. |
| Soave M, Stoddart LA, Brown A, Woolard J, Hill SJ. | Pharmacol Res Perspect. 2016 Aug 8;4(5):e00250. | |
|
1,3-Dimethyl Benzimidazolones Are Potent, Selective Inhibitors of the BRPF1 Bromodomain. |
Demont EH, Bamborough P, Chung CW, Craggs PD, Fallon D, Gordon LJ, Grandi P, Hobbs CI, Hussain J, Jones EJ, Le Gall A, Michon AM, Mitchell DJ, Prinjha RK, Roberts AD, Sheppard RJ, Watson RJ. | ACS Med Chem Lett. 2014 Sep 10;5(11):1190-5. |
| Theodoulou NH, Bamborough P, Bannister AJ, Becher I, Bit RA, Che KH, Chung CW, Dittmann A, Drewes G, Drewry DH, Gordon L, Grandi P, Leveridge M, Lindon M, Michon AM, Molnar J, Robson SC, Tomkinson NC, Kouzarides T, Prinjha RK, Humphreys PG. | J Med Chem. 2016 Feb 25;59(4):1425-39. | |
|
AZD5153: A Novel Bivalent BET Bromodomain Inhibitor Highly Active against Hematologic Malignancies. |
Rhyasen GW, Hattersley MM, Yao Y, Dulak A, Wang W, Petteruti P, Dale IL, Boiko S, Cheung T, Zhang J, Wen S, Castriotta L, Lawson D, Collins M, Bao L, Ahdesmaki MJ, Walker G, O’Connor G, Yeh TC, Rabow AA, Dry JR, Reimer C, Lyne P, Mills GB, Fawell SE, Waring MJ, Zinda M, Clark E, Chen H. | Mol Cancer Ther. 2016 Nov;15(11):2563-2574. |
|
Potent and selective bivalent inhibitors of BET bromodomains. |
Waring MJ, Chen H, Rabow AA, Walker G, Bobby R, Boiko S, Bradbury RH, Callis R, Clark E, Dale I, Daniels DL, Dulak A, Flavell L, Holdgate G, Jowitt TA, Kikhney A, McAlister M, Méndez J, Ogg D, Patel J, Petteruti P, Robb GR, Robers MB, Saif S, Stratton N, Svergun DI, Wang W, Whittaker D, Wilson DM, Yao Y. | Nat Chem Biol. 2016 Dec;12(12):1097-1104. |
| Thean D, Ebo JS, Luxton T, Lee XC, Yuen TY, Ferrer FJ, Johannes CW, Lane DP, Brown CJ. | Sci Rep. 2017 May 11;7(1):1763. | |
|
The tumour suppressor APC promotes HIV-1 assembly via interaction with Gag precursor protein |
Miyakawa K, Nishi M, Matsunaga S, Okayama A, Anraku M, Kudoh A, Hirano H, Kimura H, Morikawa Y, Yamamoto N, Ono A, Ryo A. | Nature Communications 2017 Jan 30;8:14259 |
|
PRDM14 directly interacts with heat shock proteins HSP90α and glucose-regulated protein 78. |
Moriya C, Taniguchi H, Nagatoishi S, Igarashi H, Tsumoto K, Imai K | Cancer Sci. 2018 Feb;109(2):373-383 |
| Alcobia DC, Ziegler AI, Kondrashov A, Comeo E, Mistry S, Kellam B, Chang 2, Woolard J, Hill SJ, Sloan EK | iScience 2018 Aug 31;6:280-288. doi: 10.1016/j.isci.2018.08.006. |
NanoBiT
※ 論文タイトルをクリックするとPubMedに移動します| タイトル | 著者 | ジャーナル名 |
|---|---|---|
|
NanoLuc Complementation Reporter Optimized for Accurate Measurement of Protein Interactions in Cells |
Dixon AS, Schwinn MK, Hall MP, Zimmerman K, Otto P, Lubben TH, Butler BL, Binkowski BF, Machleidt T, Kirkland TA, Wood MG, Eggers CT, Encell LP, Wood KV. | ACS Chem Biol. 2016 Feb 19;11(2):400-8. |
|
Self-Assembling NanoLuc Luciferase Fragments as Probes for Protein Aggregation in Living Cells |
Zhao J, Nelson TJ, Vu Q, Truong T, Stains CI. | ACS Chem Biol. 2016 Jan 15;11(1):132-8. |
|
SOD1 dimerization monitoring using a novel split NanoLuc, NanoBit |
Oh-Hashi K, Hirata Y, Kiuchi K. | Cell Biochem Funct. 2016 Oct;34(7):497-504. |
| Rouault AAJ, Lee AA, Sebag JA. | Biochim Biophys Acta. 2017 Dec;1864(12):2322-2329. | |
| Bodle CR, Hayes MP, O’Brien JB, Roman DL. | Anal Biochem. 2017 Apr 1;522:10-17. | |
| Koushyar S, Economides G, Zaat S, Jiang W, Bevan CL, Dart DA. | Oncogenesis. 2017 May 15;6(5):e333. | |
|
An enzyme-mediated protein-fragment complementation assay for substrate screening of sortase A |
Li N, Yu Z, Ji Q, Sun J, Liu X, Du M, Zhang W. | Biochem Biophys Res Commun. 2017 Apr 29;486(2):257-263. |
|
TDP-43 self-interaction is modulated by redox-active compounds Auranofin, Chelerythrine and Riluzole |
Oberstadt M, Stieler J, Simpong DL, Römuß U, Urban N, Schaefer M, Arendt T, Holzer M. | Sci Rep. 2018 Feb 2;8(1):2248. |
|
Self-assembly of FUS through its low-complexity domain contributes to neurodegeneration |
Matsumoto T, Matsukawa K, Watanabe N, Kishino Y, Kunugi H, Ihara R, Wakabayashi T, Hashimoto T, Iwatsubo T. | Hum Mol Genet. 2018 Apr 15;27(8):1353-1365. |
| Omachi K, Kamura M, Teramoto K, Kojima H, Yokota T, Kaseda S, Kuwazuru J, Fukuda R, Koyama K, Matsuyama S, Motomura K, Shuto T, Suico M, Kai H. | Cell Chem Biol. 2018 May 17;25(5):634-643.e4. | |
| Norisada J, Fujimura K, Amaya F, Kohno H, Hirata Y, Oh-Hashi K, | Mol Biotechnol. 2018 Jun 6. doi: 10.1007/s12033-018-0092-5. [Epub ahead of print] |